늦깎이 공대생의 인공지능 연구실
[논문프리뷰] 잠재 확산을 통한 생성용 단백질 접힘 모델의 재조명(Repurposing Protein Folding Models for Generation with Latent Diffusion) 본문
[논문프리뷰] 잠재 확산을 통한 생성용 단백질 접힘 모델의 재조명(Repurposing Protein Folding Models for Generation with Latent Diffusion)
Justin T. 2025. 4. 27. 11:47
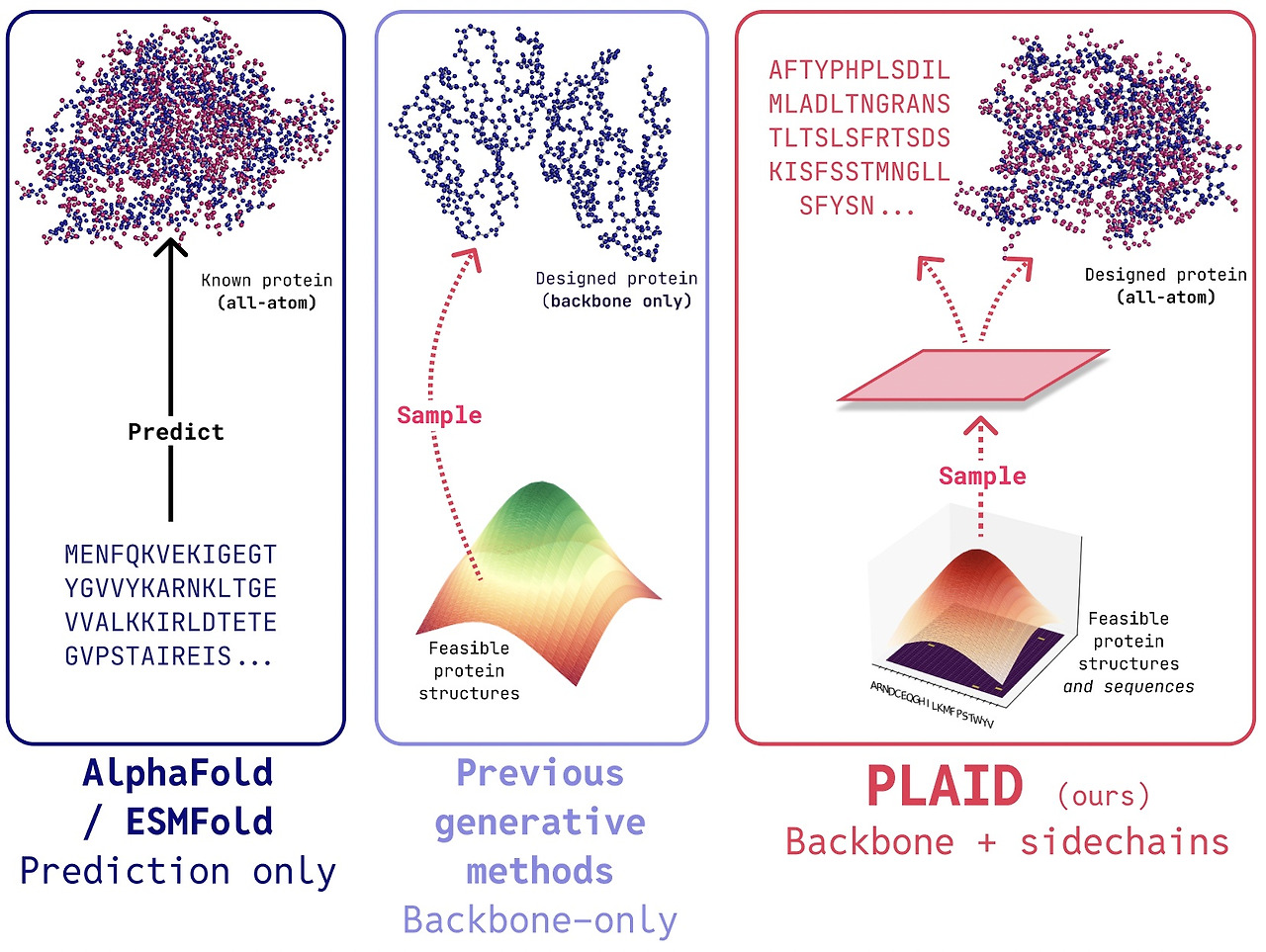
AlphaFold2의 2024년 노벨상 수상은 생물학에서 AI의 역할이 인정받는 중요한 순간이었습니다. 단백질 접힘 모델 이후 다음으로 등장하게 될 기술은 무엇이 될까요?
PLAID에서는 단백질 접힘 모델의 잠재 공간에서 샘플링을 학습하여 새로운 단백질을 생성하는 방법을 개발합니다. 구성 기능과 유기체 프롬프트를 수용할 수 있으며 구조 데이터베이스보다 2~4배 더 큰 서열 데이터베이스로 학습할 수 있습니다. 이전의 많은 단백질 구조 생성 모델과 달리 PLAID는 이산 서열과 연속적인 전체 원자 구조 좌표를 동시에 생성하는 멀티모달 공동 생성 문제 설정을 해결할 수 있습니다.
구조 예측에서 실제 약물 설계까지
최근의 연구는 확산 모델의 단백질 생성 능력에 대한 가능성을 보여주었지만, 이전 모델의 한계가 여전히 존재하여 실제 응용 분야에서는 다음과 같이 실용적이지 못했습니다
- 모든 원자 생성: 기존의 많은 생성 모델은 백본 원자만 생성합니다. 전체 원자 구조를 생성하고 사이드체인 원자를 배치하려면 순서를 알아야 합니다. 이로 인해 이산 및 연속 모달리티를 동시에 생성해야 하는 다중 모달리티 생성 문제가 발생합니다.
- 유기체 특이성: 인간이 사용하기 위한 단백질 생물학적 의약품은 인간의 면역 체계에 의해 파괴되지 않도록 인간 친화적일 필요가 있습니다.
- 제어 사양: 약물을 발견하고 이를 환자에게 제공하는 것은 복잡한 과정입니다. 이러한 복잡한 제약 조건을 어떻게 규정할 수 있을까요? 예를 들어, 생물학적인 문제를 해결한 후에도 정제가 약병보다 운반하기 쉽다고 판단하여 용해성에 대한 새로운 제약 조건을 추가할 수 있습니다.
“유용한” 단백질 생성
단백질을 단순히 생성하기만 하는 것은 유용한 단백질을 얻기 위해 생성을 제어하는 것만큼 유용하지 않습니다. 이를 위한 인터페이스는 어떤 모습일까요?

PLAID는 제어 사양을 위해 이 인터페이스를 미러링합니다. 궁극적인 목표는 텍스트 인터페이스를 통해 전적으로 생성을 제어하는 것이지만 여기서는 개념 증명으로 기능과 유기체라는 두 가지 축에 대한 구성 제약을 고려합니다.
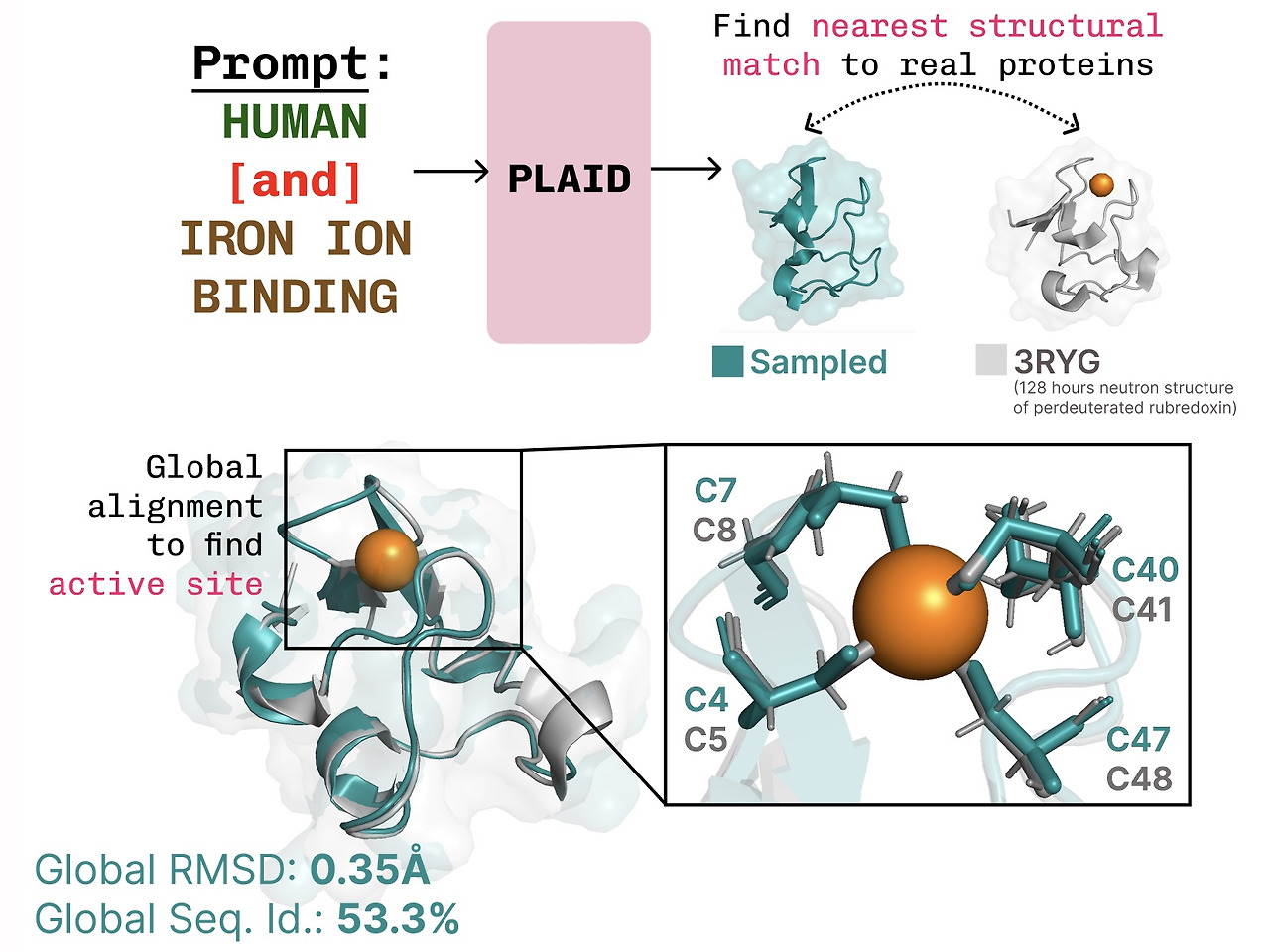
서열만 있는 학습 데이터를 사용한 학습
PLAID 모델의 또 다른 중요한 측면은 생성형 모델을 학습시키는 데 서열만 필요하다는 점입니다! 생성형 모델은 학습 데이터에 의해 정의된 데이터 분포를 학습하며, 서열 데이터베이스는 실험 구조보다 훨씬 저렴하게 얻을 수 있기 때문에 구조 데이터베이스보다 훨씬 더 큽니다.

어떻게 사용하나요?
서열 데이터만으로 구조를 생성하는 모델을 학습할 수 있는 이유는 단백질 접힘 모델의 잠재 공간에 대한 확산 모델을 학습하기 때문입니다. 그런 다음 추론 중에 유효한 단백질의 잠재 공간에서 샘플링한 후 단백질 접힘 모델에서 고정 가중치를 가져와 구조를 해독할 수 있습니다. 여기서는 검색 단계를 단백질 언어 모델로 대체하는 AlphaFold2 모델의 후속 모델인 ESMFold를 사용합니다..

이러한 방식으로 단백질 설계 작업을 위해 사전 학습된 단백질 접힘 모델의 가중치에 구조적 이해 정보를 사용할 수 있습니다. 이는 로봇 공학에서 시각-언어-행동(VLA) 모델이 인터넷 규모의 데이터로 학습된 시각-언어 모델(VLM)에 포함된 선행 정보를 활용하여 지각과 추론 및 이해 정보를 제공하는 것과 유사합니다.
단백질 접힘 모델의 잠재 공간 압축하기
이 방법을 직접 적용하는 데 따르는 작은 단점은 ESMFold의 잠재 공간(실제로 많은 트랜스포머 기반 모델의 잠재 공간)에 많은 정규화가 필요하다는 점입니다. 이 공간은 또한 매우 크기 때문에 이 임베딩을 학습하는 것은 결국 고해상도 이미지 합성에 매핑됩니다.
이를 해결하기 위해 단백질 서열과 구조의 공동 임베딩을 위한 압축 모델을 학습하는 CHEAP(Compressed Hourglass Embedding Adaptations of Proteins)도 제안합니다.

이 잠재 공간은 실제로 압축성이 매우 높다는 것을 발견했습니다. 우리가 작업하고 있는 기본 모델을 더 잘 이해하기 위해 약간의 기계론적 해석을 수행하여 전 원자 단백질 생성 모델을 만들 수 있었습니다.
다음 단계는 무엇인가요?
다음 단계는 무엇인가요?
이 작업에서는 단백질 서열과 구조 생성의 경우를 살펴보았지만, 이 방법을 적용하면 더 풍부한 양식에서 덜 풍부한 양식으로 추정할 수 있는 예측기가 있는 모든 양식에 대해 다중 양식 생성을 수행할 수 있습니다. 단백질의 서열 대 구조 예측기가 점점 더 복잡한 시스템을 다루기 시작함에 따라(예: AlphaFold3는 핵산 및 분자 리간드와 복합된 단백질도 예측할 수 있음), 동일한 방법을 사용하여 더 복잡한 시스템에 대해 다중 모드 생성을 수행하는 것을 쉽게 상상할 수 있습니다.
참고자료





